[파이썬 머신러닝 완벽 가이드 (권철민)]

2장. 사이킷런으로 시작하는 머신러닝 - Chapter 03 평가
[분류의 성능평가 지표]
• 정확도(accuracy)
• 오차 행렬(confusion matrix)
• 정밀도(precision)
• 재현율(recall)
• F1 스코어
• ROC AUC
1. F1 스코어(F1 Score)
F1 score는 정밀도와 재현율을 결합한 지표입니다., 정밀도와 재현율이 어느 한쪽으로 치우치지 않는 수치를 나타낼 때 상대적으로 높은 값을 가집니다.
F1 스코어는 정밀도(Precision)와 재현율(Recall)의 조화 평균(Harmonic Mean)을 나타내는 지표입니다. 조화 평균은 정밀도와 재현율의 값이 둘 다 높아야 전체적으로 높은 값을 얻을 수 있기 때문에, 두 지표 모두 중요하게 여겨질 때 유용합니다.
$$
F1 = { 2 \over {1 \over recall} + {1 \over precision}} = 2*{ precision * recall\over precision+ recall }
$$
F1 스코어는 0에서 1 사이의 값을 가지며, 1에 가까울수록 모델의 성능이 뛰어난 것을 나타냅니다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
|
titanic = pd.read_csv('./drive/MyDrive/data/titanic_train.csv')
y_titanic = titanic.Survived
X_titanic = titanic.drop('Survived', axis=1)
X_titanic = transform_features(X_titanic)
X_train, X_test, y_train, y_test = train_test_split(X_titanic, y_titanic, test_size=0.2, random_state=2)
lr_clf = LogisticRegression(max_iter=5000, random_state=2)
lr_clf.fit(X_train, y_train)
pred = lr_clf.predict(X_test)
get_clf_eval(y_test, pred)
pred_proba = lr_clf.predict_proba(X_test)
#print(pred_proba)
f1 = f1_score(y_test, pred)
print('F1 스코어: {0:.4f}'.format(f1))
|
cs |

hreshold 변화에 따른 F1 스코어를 구해보았고, 이전의 get_clf_eval() 함수에 F1 스코어를 구하는 로직을 추가하였다.
그리고 이전의 get_eval_binar() 함수를 이용해 0.4 ~ 0.6 별로 평가 지표를 구해보았다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
|
def get_clf_eval(y_test , pred):
confusion = confusion_matrix( y_test, pred)
accuracy = accuracy_score(y_test , pred)
precision = precision_score(y_test , pred)
recall = recall_score(y_test , pred)
# F1 스코어 추가
f1 = f1_score(y_test,pred)
print('오차 행렬')
print(f'TN {confusion[0][0]}\t/ FP {confusion[0][1]}')
print(f'FN {confusion[1][0]}\t/ TP {confusion[1][1]}')
# f1 score print 추가
print('정확도: {0:.4f}, 정밀도: {1:.4f}, 재현율: {2:.4f}, F1:{3:.4f}'.format(accuracy, precision, recall, f1))
def get_eval_binar(thresholds, y_tests, pred_probas):
binar = Binarizer(threshold=thresholds)
y_binar = binar.fit_transform(pred_probas)
print('====', 'thresholds', ' : ', thresholds)
get_clf_eval(y_tests, y_binar[:, 1] )
pred_proba = lr_clf.predict_proba(X_test)
thresholds = [0.4 , 0.45 , 0.50 , 0.55 , 0.60]
for i in thresholds:
get_eval_binar(i, y_test, pred_proba)
|
cs |

제공된 결과를 보면, 각 임곗값에 따라 오차 행렬과 성능 지표가 어떻게 변화하는지 볼 수 있습니다. 임곗값을 증가시킬 때마다 다음과 같은 경향을 볼 수 있습니다:
• 정밀도는 증가합니다. 이는 임곗값을 높임으로써 모델이 양성으로 예측하는 경우가 더 엄격해져 실제 양성인 경우의 비율이 높아지기 때문
• 재현율은 감소합니다. 이는 임곗값이 높아짐에 따라 실제 양성을 양성으로 예측하지 못하는 경우가 많아지기 때문
• F1 스코어는 정밀도와 재현율의 조화 평균을 나타내므로, 두 값 사이의 균형을 나타내는 지표로, 최적의 임곗값은 정밀도와 재현율이 어느 정도 균형을 이루는 지점에서 높은 F1 스코어를 보입니다.
이러한 결과는 모델이 특정 클래스를 어떻게 예측하고 있는지, 그리고 정밀도와 재현율 사이의 균형을 어떻게 설정해야 하는지에 대한 통찰력을 제공합니다. 예를 들어, 질병을 진단하는 모델이라면, 재현율을 더 중요시 할 수 있고, 스팸 메일 필터링과 같은 경우에는 정밀도가 더 중요할 수 있습니다.
위의 결과에서 볼 수 있듯이, 임곗값을 높이면 정밀도는 상승하지만 재현율이 떨어져 F1 스코어도 낮아질 수 있습니다. 반대로 임곗값을 너무 낮추면 재현율은 높아지지만 정밀도가 감소하여 다시 F1 스코어가 낮아질 수 있습니다. 즉, 임곗값을 조정함으로써 모델의 F1 스코어를 최적화할 수 있습니다.
제시된 값들 중에서 임곗값이 0.5일 때 F1 스코어가 0.7183으로 상대적으로 높게 나타나는 것을 볼 수 있습니다. 이는 해당 임곗값에서 모델의 정밀도와 재현율 사이의 균형이 상대적으로 좋음을 나타냅니다. F1 스코어는 모델이 양성 클래스를 어떻게 예측하는지에 대한 종합적인 성능을 나타내므로, 이를 기준으로 임곗값을 선택할 수 있습니다.
2. ROC곡선과 AUC
ROC 곡선과 이에 기반한 AUC 스코어는 이진 분류의 예측 성능 측정에서 중요하게 사용되는 지표입니다.
ROC 곡선(Receiver Operation Characteristic Curve)는 일반적으로 의학 분야에서 많이 사용되지만, ML의 이진 분류 모델의 예측 성능을 판단하는 중요한 평가 지표이기도 하다.
ROC 곡선은 FPR(Flase Positive Rate)가 변할 때 TPR(True Positive Rate)가 어떻게 변하는지를 나타내는 곡선이다.
FPR을 X 축으로, TPR을 Y축으로 잡으면 FPR변화에 따른 TPR 변화가 곡선 형태로 나타납니다.
여기서 TPR은 TP / (FN + TP)로 재현율을 의미하며 민감도라고도 불린다.
그리고 민감도에 대응하는 지표로는 TNR(True Negative Rate)이라고 부리는 특이성(Specificity)이 이다.
• 민감도(TPR)는 실제값 Positive(양성)가 정확히 예측돼야 하는 수준을 나타낸다.
(질병이 있는 사람은 질병이 있는 것으로 양성 판정)
• 특이성(TNR)은 실제값 Negative(음성)가 정확히 예측돼야 하는 수준을 나타낸다.
(질병이 없는 건강한 사람은 질병이 없는 것으로 음성 판정)
- TNR은 TN / (FP + TN)으로 구할 수 있고,
- FPR은 FP / (FP + TN)이므로 - 1-TNR 또는 1-특이성으로 표현된다. / FPR = FP / (FP + TN) = 1 - 특이성
• FPR = False positive rate
• TPR = True positive rate
1. TPR = TP / (TP+FN) = recall
▶ FN = 0, TPR = 1
2. TNR = TN / (TN+FP) = specificity
▶ FP = 0, TNR = 1
3. FPR = FP / (TN+FP) = 1- TNR
▶ FP = 0, FPR = 0
$$
{TN+FP \over TN+FP} - {TN \over TN+FP} = 1 - TNR
$$

위 이미지는 ROC 곡선의 예이다.
가운데 직선은 ROC 곡선의 최저값이고, ROC 곡선이 가운데 직선에 가까울수록 성능이 떨어지는 것이며, 멀어질수록 성능이 뛰어난 것이다.
ROC 곡선은 FPR을 0부터 1까지 변화시키면서 TPR의 변화값을 구합니다.. 그리고 FPR을 0부터 1까지 변화시키는 방법은 분류 결정 threshold를 변화시키면 된다. threshold는 Positive 예측값을 결정하는 확률의 기준이기 때문에 FPR을 0으로 만드려면 threshold는 1로 지정하면 된다.
FPR은 FP / (FP + TN)이기 때문에 Positive로 예측하는 경우가 없도록 FP가 0이 되면 FPR도 0이 된다.
반대로 FPR을 1로 만드려면 TN을 0으로 만들면 되기 때문에 Negative로 예측하는 경우가 없도록 threshold를 0으로 만들면 된다.
따라서 ROC 곡선은 threshold 값을 1부터 0까지 변화시키며 FPR을 구하고, FPR 값의 변화에 따른 TPR 값을 구하는 곡선이다.
사이킷런은 ROC 곡선을 구하기 위해 roc_curve() API를 제공한다.
사용법은 precesion_recall_curve()와 유사하다. 반환값이 FPR, TPR, 임계값으로 구성되어 있을 뿐이다.,
| 입력 파라미터 | y_true : 실제 크래스 값 array (array shape=[데이터 건수]) y_score : predict_proba()의 반환 값 array에서 Positive 커럼의 예측 확률이 보통 사용됨. (array_shape=[n_samples]) |
| 반환 값 | fpr : fpr 값을 array로 반환 tpr : tpr 값을 array로 반환 thresholds : threshold 값 array |
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
|
from sklearn.metrics import roc_curve
#roc_curve 입력값 : y_test, 1(=positive)의 확률
#roc_curve 출력값 : fpr, tpr, thresholds
# 레이블 값이 1일때의 예측 확률을 추출
pred_proba_class1 = lr_clf.predict_proba(X_test)[:, 1]
fprs , tprs , thresholds = roc_curve(y_test, pred_proba_class1)
# 반환된 임곗값 배열에서 샘플로 데이터를 추출하되, 임곗값을 5 Step으로 추출.
# thresholds[0]은 max(예측확률)+1로 임의 설정됨. 이를 제외하기 위해 np.arange는 1부터 시작
thr_index = np.arange(1, thresholds.shape[0], 5)
print('샘플 추출을 위한 임곗값 배열의 index:', thr_index)
print('샘플 index로 추출한 임곗값: ', np.round(thresholds[thr_index], 2))
# 5 step 단위로 추출된 임계값에 따른 FPR, TPR 값
print('샘플 임곗값별 FPR: ', np.round(fprs[thr_index], 3))
print('샘플 임곗값별 TPR: ', np.round(tprs[thr_index], 3))
|
cs |

roc_curve()의 결과 살펴보면 임곗값이 1에 가까운 값에서 점점 작아지면서 FPR이 점점 커집니다. 그리고 FPR이 조금씩 커질떄 TPR은 가파르게 커짐을 알 수 있습니다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
|
pred_proba_class1 = lr_clf.predict_proba(X_test)[:, 1]
fprs, tprs, thresholds = roc_curve(y_test, pred_proba_class1)
plt.figure(figsize=(6,6))
plt.plot(fprs, tprs, label = "ROC")
plt.plot([0,1],[0,1], 'k--', label = 'Random')
plt.xlabel('FPR',fontsize=25)
plt.ylabel('TPR',fontsize=25)
plt.tick_params(length=10, labelsize=20)
plt.title('ROC Curve',fontsize=25)
plt.tight_layout()
plt.grid()
plt.legend(loc='lower right')
|
cs |

ROC곡선 자체는 FPR과 TPR의 변화 값을 보는데 이용하며, 분류의 성증 지표로 사용되는 것은 ROC 곡선 면적에 기반한 AUC값으로 결정합니다.
AUC(Area Under Curve) 값은 AOC 곡선 밑의 면적을 구한 것으로서 일반적으로 1에 가까울수록 좋은 수치입니다. AUC가 커지려면 FPR이 작은 상태에서 얼마나 큰 TPR을 얻을 수 있느냐가 관건입니다.
|
1
2
3
4
5
|
from sklearn.metrics import roc_auc_score
pred_proba = lr_clf.predict_proba(X_test)[:,1]
roc_score = roc_auc_score(y_test, pred_proba)
print('ROC AUC 값: {0: .4f}'.format(roc_score))
|
cs |

• get_clf_eval() 함수에 ROC AUC값 추가
|
1
2
3
4
5
6
7
8
9
10
11
12
13
|
def get_clf_eval(y_test , pred, pred_probas=None):
confusion = confusion_matrix( y_test, pred)
accuracy = accuracy_score(y_test , pred)
precision = precision_score(y_test , pred)
recall = recall_score(y_test , pred)
f1 = f1_score(y_test,pred)
# ROC-AUC 추가
roc_auc = roc_auc_score(y_test, pred_probas)
print('오차 행렬')
print(f'TN {confusion[0][0]}\t/ FP {confusion[0][1]}')
print(f'FN {confusion[1][0]}\t/ TP {confusion[1][1]}')
# ROC - AUC 추가
# None 값을 문자열로 처리하도록 수정 roc_auc_str = 'None' if roc_aucs is None else f'{roc_aucs:.4f}' print('정확도: {0:.4f}, 정밀도: {1:.4f}, 재현율: {2:.4f}, F1:{3:.4f},AUC:{4:.4f}'.format(accuracy, precision, recall, f1, roc_auc_str))
|
cs |
파이썬 sklearn 유방암 데이터 (load_breast_cancer)
1. 데이터 불러오기 / 데이터 확인하기


- 데이터 Label 확인

2. 데이터프레임으로 확인하기




문제 풀어보기
[조건]
1. 모델 LogisticRegression(max_iter=5000)
2. accuracy, precision, recall, f1, roc_auc 구하기
3. DecisionTreeClassifier
4. RandomForestClassifier
|

제시된 결과는 로지스틱 회귀(Logistic Regression), 결정 트리(Decision Tree), 그리고 랜덤 포레스트(Random Forest) 세 가지 분류 모델을 이용하여 특정 데이터셋에 대한 예측 성능을 평가한 것입니다. 평가 지표로는 정확도(Accuracy), 정밀도(Precision), 재현율(Recall), F1 스코어(F1 Score), 그리고 AUC(Area Under the Curve) 값이 사용되었습니다.
[결과 해석]
• 로지스틱 회귀(Logistic Regression):
- 정확도: 91.81%, 정밀도: 93.16%, 재현율: 94.78%, F1 스코어: 93.97%, AUC: 98.35%
• 결정 트리(Decision Tree):
- 정확도: 91.23%, 정밀도: 92.37%, 재현율: 94.78%, F1 스코어: 93.56%, AUC: 89.36%
• 랜덤 포레스트(Random Forest):
정확도: 94.15%, 정밀도: 94.87%, 재현율: 96.52%, F1 스코어: 95.69%, AUC: 98.66%
[해석 포인트]
▶ 정확도(Accuracy): 모델이 전체 샘플 중 얼마나 많은 샘플을 정확하게 분류했는지를 나타냅니다. 모든 모델이 비교적 높은 정확도를 보였으며, 특히 랜덤 포레스트가 가장 높은 정확도를 보였습니다.
▶ 정밀도(Precision): 모델이 양성으로 분류한 것 중 실제 양성인 것의 비율입니다. 모든 모델이 90% 이상의 정밀도를 보였으며, 이는 모델이 상당히 신뢰할 수 있는 양성 예측을 제공한다는 것을 의미합니다.
▶ 재현율(Recall): 실제 양성 중 모델이 양성으로 올바르게 예측한 것의 비율입니다. 모든 모델이 비슷한 재현율을 보였으며, 특히 랜덤 포레스트가 가장 높은 재현율을 보였습니다.
▶ F1 스코어(F1 Score): 정밀도와 재현율의 조화 평균입니다. 높은 F1 스코어는 모델이 정밀도와 재현율 사이에 좋은 균형을 이루고 있음을 의미합니다. 모든 모델이 높은 F1 스코어를 보였으며, 랜덤 포레스트가 가장 높았습니다.
▶ AUC(Area Under the Curve): ROC 곡선 아래의 면적으로, 모델이 임의의 양성 샘플보다 임의의 음성 샘플을 더 높은 확률로 예측하는 능력을 나타냅니다. 랜덤 포레스트가 가장 높은 AUC 값을 보였으며, 이는 모델의 전반적인 성능이 우수함을 의미합니다.
종합
위 결과를 종합해보면, 랜덤 포레스트 모델이 이 데이터셋에 대해 가장 뛰어난 성능을 보였음을 알 수 있습니다. 랜덤 포레스트는 높은 정확도, 정밀도, 재현율, F1 스코어, 그리고 AUC 값을 달성하였습니다. 이는 랜덤 포레스트가 복잡한 데이터셋을 잘 처리하며, 다른 두 모델에 비해 우수한 일반화 성능을 가지고 있음을 시사합니다.
파마 인디언 당뇨병 예측 문제
피마 인디언 당뇨병 dataset을 이용하여 당뇨병 여부를 판단하는 ML 예측 모델을 만들고 평가 지표들을 적용
[피마 인디언 당뇨병 dataset의 피쳐 구성]
- Pregnancies : 임신 횟수
- Glucose : 포도당 부하 검사 수치
- BloodPressure : 혈압(nm Hg)
- SkinThickness : 팔 삼두근 뒤쪽의 피하지방 측정값(nm)
- Insulin : 혈청 인슐린(mu U/ml)
- BMI : 체질량지수(체중(kg)/(키(m))^2)
- DiabetesPedigreeFunction : 당뇨 내력 가중치 값
- Age : 나이
- Outcome : Class 결정 값(0 or 1)
1. 데이터 세트 확인
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
|
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score, precision_score, recall_score, roc_auc_score
from sklearn.metrics import f1_score, confusion_matrix, precision_recall_curve, roc_curve
from sklearn.preprocessing import StandardScaler, Binarizer
from sklearn.linear_model import LogisticRegression
diabetes_data = pd.read_csv('./drive/MyDrive/data/diabetes.csv')
print(diabetes_data['Outcome'].value_counts())
diabetes_data.head(5)
|
cs |

- 아상치 확인
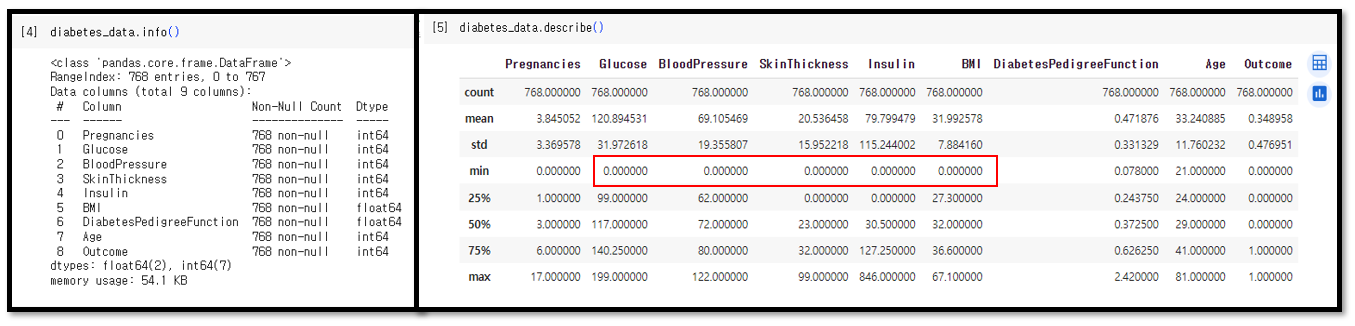
min() 값이 0인 것이 많이 눈에 띈다. 예로들어 Glucose는 포도당 수치인데 이것이 0인 것은 말이 안된다.

|
1
2
3
4
5
6
7
8
9
|
zero_features = ['Glucose', 'BloodPressure', 'SkinThickness','Insulin','BMI']
def zero_check(zero_features):
for feature in zero_features:
zero_count = diabetes[diabetes[feature]==0][feature].count()
total_count = diabetes[feature].count()
zero_rate = np.round((zero_count/total_count)*100,2)
print(feature,' : ', zero_rate,'%')
zero_check(zero_features)
|
cs |

min() 값이 0으로 되어 있는 피처에 대해 0 값의 건수 및 전체 데이터 건수 대비 몇퍼센트의 비율로 존재하는지 확인
2. 데이터 변환/ 0의 데이터 평균으로 대체

SkinThickness와 Insulin의 값은 각각 전체의 29.56%, 48.7%로 상당히 많습니다.
전체 데이터 건수가 많지 않기 떄문에 이들을 삭제하는 것이 아닌 0의 값을 평균값으로 대체시켜줍니다.
3. 데이터 피처 스케일링
0 값을 평균값으로 대체한 데이터 세트에 피처 스케일링을 적용해 변환해보자. 일반적으로 로지스틱 회귀의 경우 숫자 데이터에 스케일링을 적용하는 것이 좋다.
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
|
X = diabetes_data.iloc[:, :-1]
y = diabetes_data.iloc[:, -1]
# StandardScaler 클래스를 이용해 피처 데이터 세트에 일괄적으로 스케일링 적용
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=156, stratify=y)
lr_clf = LogisticRegression()
lr_clf.fit(X_train, y_train)
pred = lr_clf.predict(X_test)
pred_proba = lr_clf.predict_proba(X_test)[:, 1]
get_clf_eval(y_test, pred, pred_proba)
|
cs |

|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
|
def fit_predict_eval(X_train, y_train, X_test, y_test):
lr_clf = LogisticRegression(max_iter=5000)
lr_clf.fit(X_train, y_train)
pred = lr_clf.predict(X_test)
pred_proba = lr_clf.predict_proba(X_test)[:, 1]
get_clf_eval(y_test, pred, pred_proba)
from sklearn.preprocessing import StandardScaler, MinMaxScaler
#MinMaxScaler() 사용
mms = MinMaxScaler()
X_mms = mms.fit_transform(X)
X_train, X_test, y_train, y_test = train_test_split(X_mms, y_label, test_size=0.2, random_state= 156, stratify=y_label)
fit_predict_eval(X_train, y_train, X_test, y_test)
# StandardScaler() 사용
ss = StandardScaler()
X_ss = ss.fit_transform(X)
X_train, X_test, y_train, y_test = train_test_split(X_ss, y_label, test_size=0.2, random_state= 156, stratify=y_label)
fit_predict_eval(X_train, y_train, X_test, y_test)
|
cs |

종합 해석:
두 모델은 매우 유사한 정확도를 보이고 있지만, 미묘한 차이가 있습니다. 첫 번째 모델은 조금 더 높은 정밀도를 보였지만 재현율이 낮았고, 두 번째 모델은 재현율이 더 높지만 정밀도가 낮았습니다. 이러한 차이는 F1 스코어에서도 반영되어 두 번째 모델이 더 균형 잡힌 성능을 보이고 있습니다.
재현율이 중요한 상황에서는 두 번째 모델이 더 선호될 수 있고, 정밀도를 더 중요시하는 상황에서는 첫 번째 모델이 더 적합할 수 있습니다. AUC 점수는 두 모델 모두 비교적 높아, 일반적으로 두 모델 모두 양호한 예측 성능을 가지고 있다고 볼 수 있습니다.
[정리]
지금까지 분류에 사용되는 정확도, 오차행렬, 정밀도, 재현율, F1 스코어, ROC- AUC와 같은 성능 평가 지표를 살펴봤습니다. 특히 이진 분류의 레이블 값이 불균형하게 분포될 경우(즉 0이 매우 많고, 1이 매우적을 경우 또는 반대 상황) 단순히 예측 결과와 실제 결과가 일치하는 지표인 정확도만으로는 머신러닝 모델의 예측 성능을 평가 할 수 없습니다.
오차행열은 Negative와 Positive 값을 ㅏ지는 실제 클래스 값과 예측 클래스 값이 True와 False에 따라 TN,FP,FN,TP로 매핑 되는 4분명 행렬을 기반으로 예측 성능을 평가합니다. 정확도, 재현율, 정밀도 수치는 이들을 다양하게 결합해 만들어지며, 이를 통해 분류 모델 예측 성능의 오류가 어떠한 모습으로 발생하는지 알 수 있는 것입니다.
정밀도(Precesion)와 재현율(Recall)은 Positive데이터 세트의 예측 성능에 좀 더 초점을 맞춘 평가 지표입니다. 특히 재현율이 상대적으로 더 중요한 지표인 경우(예- 암 양성 예측 모델)는 Positive 양성인 데이터 예측을 Negative로 잘못 판단하게 되면 업무상 큰 영향이 발생하는 경우입니다.
분류하려는 업무의 특성상 정밀도 또는 재현율이 특별히 강조돼야 할 경우 분류의 결정 임곗값(Threshold)을 조정해 정밀도 또는 재현율의 수치를 높이는 방법에 대해서 배웠습니다.
F1 스코어는 정밀도와 재현율을 결합한 평가 지표이며, 정밀도와 재현율이 어느 한 쪽으로 치우치지 않을때 높은 지표 값을 가지게 됩니다.
ROC -AUC는 일반적으로 이진 분류의 성능 평가를 위해 가장 많이 사용되는 지표입니다. AUC(Area under curve) 값은 ROC 고건 밑의 면적을 구한 것으로써 일반적으로 1에 가까울수록 좋은 수치입니다.
'데이터 공부 > 머신러닝 공부' 카테고리의 다른 글
| 파이썬 머신러닝 완벽 가이드 (권철민) - 분류 - ① 결정트리 (0) | 2024.05.02 |
|---|---|
| 통계적 머신러닝 - Sklearn /데이터 인코딩 (0) | 2024.04.18 |
| 통계적 머신러닝 - Sklearn / 교차분석/ KFold / cross_val_score (0) | 2024.04.17 |
| 01. 머신러닝(marchine learning)이란? (1) | 2024.04.15 |



